Ajou News
NEW 아주대·가톨릭대 연구팀, mRNA 백신·치료제 효용 높일 新 분석 기술 개발
- 2025-07-21
- 423

아주대·가톨릭대 공동 연구팀이 mRNA 치료제의 핵심 구조로 그동안 정확한 분석이 어려웠던 ‘poly(A) 꼬리’의 길이를 정확하게 분석할 수 있는 기술을 개발했다. 이에 기존 치료제의 한계를 넘어 감염병과 암, 유전질환 등의 치료에 기여할 것으로 주목되는 mRNA 기반 백신 및 치료제의 품질관리 기술로 활용될 수 있을 전망이다.
박대찬 아주대 교수(첨단바이오융합대학·대학원 분자과학기술학과, 사진)와 남재환 가톨릭대 교수(의생명과학과) 공동 연구팀은 mRNA 치료제의 핵심 구조인 poly(A) 꼬리(poly(A)denylation tail)의 길이를 정확하게 정량 분석할 수 있는 새로운 기술을 개발했다고 밝혔다.
해당 연구 결과는 ‘3AIM-seq: 체외 전사된 mRNA의 3’ 말단 poly(A) 꼬리 시퀀싱을 활용한 mRNA 치료제의 품질평가(3AIM-seq: Quality assessment of mRNA therapeutics using sequencing for 3′ poly(A) tails of in vitro-transcribed mRNA)’라는 제목으로 세포・유전자 치료 및 RNA 기반 치료제 분야의 저명 학술지인 <몰레큘러 테라피(Molecular Therapy)>에 6월28일 온라인 게재됐다.
이번 연구에는 아주대 박대찬 교수와 가톨릭대 남재환 교수가 공동 교신저자로 참여했고, 아주대 서진아 석사 졸업생(현 넥스아이), 가톨릭대 박효정 박사(현 한국보건산업진흥원 질병청), 가톨릭대 오아영 석사 졸업생(현 가톨릭대 융합과학기술연구소)이 공동 제1저자로 참여했다. 서울대 장혜식 교수 연구팀과 한국생명공학연구원 김천아 박사 연구팀은 공동 저자로 참여했다.
mRNA(메신저 리보핵산)는 세포 안에서 단백질을 만드는 역할을 하며, mRNA 치료제는 질병을 일으키는 단백질 부족을 보충하거나, 특정 질병에 대한 면역 반응을 유도하는 단백질을 만들도록 세포에 지시하는 방식으로 작동한다. mRNA 기술은 감염병 백신 개발과 암, 유전질환과 자가면역질환 치료 등에의 활용을 위해 널리 연구되고 있다. 실제로 코로나19 감염병의 전 세계적 확산에 대응해, mRNA 기술이 코로나19 백신 개발에 활용되어 전례 없이 빠르게 실제 접종까지 이어진 바 있다. 기존 백신은 개발에만 10년 이상 소요되고, 복잡한 생산 공정과 고도의 시설이 필요했던 반면, 코로나19 mRNA 백신의 경우 비교적 단순한 생산 공정으로 제조가 가능해, 동물 실험과 임상 시험 등에 8개월 정도가 소요되었던 것. 코로나19 백신은 mRNA 기술을 이용한 의약품 중 최초로 인체에 대한 대규모 접종 승인을 받은 사례다.
공동 연구팀이 주목한 poly(A) 꼬리(poly(A)denylation tail)는 mRNA 치료제의 핵심 구조다. poly(A) 꼬리란 mRNA의 3’ 말단에 위치한 아데닌 염기의 반복 서열 구조로, mRNA의 안정성 유지와 단백질 발현 효율 조절에 중요한 역할을 한다. 이에 mRNA 약물의 유효성과 안정성에 직접적인 영향을 미친다.
때문에 mRNA 치료제 개발 및 제조 공정을 위해, poly(A) 꼬리의 길이와 구조를 정밀하게 측정하고 품질을 관리하는 것은 매우 중요하다. 그러나 poly(A) 꼬리는 수십~수백 개의 동일 염기(아데닌)가 반복되는 동종중합체(homopolymer) 구조로 되어 있어, 기존 시퀀싱 기술로는 삽입・삭제 오류(indel error)가 빈번히 발생한다. 이에 정량적 평가에 한계가 있었다. 특히 최근 mRNA 치료제 poly(A) 구간의 균일성을 개선하기 위해 중간에 짧은 비동종중합체의 링커(linker) 서열을 삽입하는 방식이 많이 활용되고 있어, 더욱 복잡한 구조 분석이 요구되는 상황이었다.
이에 공동 연구팀은 치료제로 활용 가능한 구조를 갖춘 체외 전사 mRNA(IVT mRNA) 4종과 표준물질로 사용할 합성 단일 가닥 DNA(ssDNA) 올리고뉴클레오타이드를 설계 및 제작하고, 이를 기반으로 새로운 시퀀싱 분석법인 3AIM-seq를 개발했다.
연구팀은 poly(A) 꼬리 길이와 구조의 정확한 평가가 mRNA 치료제의 안정성 및 발현 효율과 직결된다는 점에 주목해, 링커 구조 포함 여부 등 poly(A) 꼬리에 대해 구조적 다양성을 갖는 4종의 체외 전사 mRNA(IVT mRNA)를 자체 제작한 뒤, 이를 3AIM-seq 분석법 성능 검증에 활용했다.
또한 차세대 유전자 분석 대표 기술인 일루미나(Illumina) 플랫폼을 이용, poly(A) 꼬리 길이를 정밀하게 산출할 수 있는 생명정보학 알고리즘을 함께 개발했다. 이를 통해 PCR 증폭 과정에서 발생할 수 있는 오차를 최소화하고, 길이 예측을 매우 정밀하게(±5 nt 정밀도) 수행할 수 있을 뿐 아니라, 링커 삽입 여부에 따라 구분되는 복합 구조도 안정적으로 식별해낼 수 있었다. 연구팀은 여기에 표준물질로 활용한 보정 모델을 도입해 분석 정확도를 향상시킴과 동시에 실제 체외 전사 mRNA(IVT mRNA)에서 poly(A) 꼬리 길이의 이질성을 정량적으로 평가하는 데 성공했다.
이번 연구는 mRNA 치료제의 구조적 품질평가에 있어 정밀도와 재현성을 동시에 갖춘 새로운 분석법을 제시한 것으로, 관련 치료제 개발 및 제조 과정에서 poly(A) 꼬리의 길이와 구조를 동시에 평가할 수 있는 실용적 도구로 활용될 수 있음을 보여준다. 이에 앞으로 백신, 항암제 등 다양한 mRNA 기반 치료제의 품질관리 기준 수립과 규제 대응에 기여할 것으로 전망된다.
아주대 박대찬 교수는 “이번 연구성과는 mRNA 치료제의 기본 형태 가운데 분석이 가장 어렵다고 여겨져 온 ‘poly(A)’의 품질을 정량적으로 평가할 수 있는 새로운 분석 기술”이라며 “연구팀이 개발한 새로운 시퀀싱 분석법(3AIM-seq)은 앞으로 mRNA 백신 및 치료제의 품질관리를 위한 핵심 도구로 활용될 수 있을 것”이라고 말했다.
이번 공동 연구에서 아주대 박대찬 교수 연구팀은 poly(A) 꼬리 분석에 최적화된 차세대염기서열 시퀀싱 방법 및 실험 디자인과 3AIM-seq 알고리즘 개발을 맡았다. 차세대염기서열 시퀀싱(NGS; next-generation sequencing)이란 DNA 혹은 RNA의 염기서열을 기존 방법보다 대용량으로 빠르고 정확하게 읽을 수 있는 분석법으로 ▲바이오마커 연구 ▲유전자 발현 연구 등 다양한 분야에서 폭넓게 활용되고 있다. 가톨릭대 남재환 교수 연구팀은 실제 치료제로 활용 가능한 구조를 갖춘 체외 전사 mRNA(IVT mRNA)의 설계 및 생산을 담당했다.
이번 연구는 교육부 G-LAMP 사업, 과학기술정보통신부 중견연구자지원사업과 식품의약품안전처지원을 받아 수행됐다.
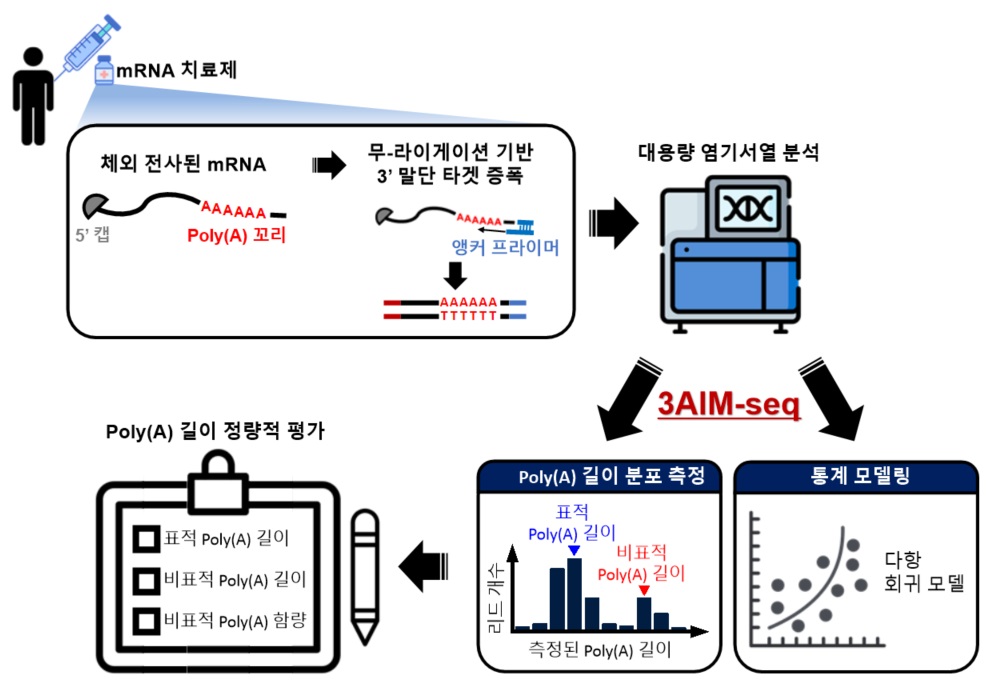
mRNA 치료제 품질의 핵심 요소인 poly(A) 꼬리의 길이를 정확하게 측정하고 정밀도를 평가하는 ‘3AIM-seq’ 분석 개요. mRNA 치료제의 poly(A) 말단을 무결합 앵커 프라이머로 증폭하고, 대용량 염기서열 분석을 수행함. 수백만 개의 염기서열을 분석해 poly(A) 길이 분포를 계산하고 통계 모델링을 수행함. 이를 통해, 설계한 표적 poly(A)와 비표적 poly(A)의 양을 계산해 mRNA 치료제 품질을 측정할 수 있음
